

9月6日,开发超灵敏测序分析技术的基因组学和分子诊断公司阅尔基因与Oxford Nanopore共同公布了一项定位于临床应用的串联扩增子纳米孔测序技术(OCEANS)。该技术能够快速、准确且经济地检测低至 0.05% 变异等位基因频率 (VAF) 的体细胞突变。这项新颖的研究将加快实现当日、低成本肿瘤检测panel的开发,适用于对时间要求高的治疗选择和复发监测。
该研究是第三代测序技术应用在单碱基变异检测上的重要尝试,相关成果发表在权威期刊Genome Biology(IF= 13.583)上。在使用临床FFPE样本比较OCEANS与主流NGS方法的一致性实验中,AUC(Area Under the Curve)区域达到99.79%~99.99%,证明二者超高的一致性。同时,ddPCR也验证了OCEANS技术在低VAF水平(0.02%~0.66%)的检测可靠性,提示OCEANS技术在MRD监测中的应用潜力。
文章发表在Genome Biology上
高通量测序正逐步渗透到癌症临床诊疗的方方面面,从伴随诊断、用药指导、预后监测到肿瘤早筛等等。
主流的以边合成边测序(SBS)为核心的NGS平台可以检测到单碱基变异(SNV),但也存在读长短、周期长的缺陷,此外高昂的实验成本也限制了其大规模推广。
相比之下,纳米孔测序(Nanopore Sequencing)具有超长读长,直接测序和无GC偏倚等优点,并且成本更低,出结果更快,可以在更精确的时间点进行癌症监控,帮助临床医生更快做出治疗决策。但目前纳米孔测序技术仍然难以实现低丰度体细胞SNV的检测。
基于此,研究团队提出了OCEANS技术,可以从FFPE样本来源的DNA中检出低于1% VAF的单碱基水平突变,并将整个流程控制在10小时以内。
为了获得尽可能长的文库序列长度,研究团队提出了随机扩增子连接(SAL)方法将短链扩增子片段串联成适合Nanopore测序平台的长reads序列,以便兼容目前临床常用的FFPE或者cfDNA样本。借助该方法将检测限提高到5%VAF,接近常规NGS方法的标准。更进一步,OCEANS引入了抑制探针置换扩增技术(BDA),能将目标区域的稀有变异富集1000倍以上。实验证实,OCEANS的检测灵敏度相比单纯纳米孔测序方法提高了100倍,测序通量提高了10倍。

图1.
Overview of Oncogene Concatenated Enriched Amplicon Nanopore Sequencing (OCEANS) 方法概述。
从肿瘤组织FFPE样本中提取的DNA样本平均长度仅为500nt,直接用于Nanopore平台会降低整体测序通量。滚环复制常被用于合成更长的待测序列,但该方法初始的环化步骤效率不高,无法满足临床样本对于灵敏度和转化率的要求。此外,复制偏差以及非双链DNA的产物形态也会影响最终结果。
为此,基于Golden Gate组装技术改造而来的SAL技术应运而生。首先,通过PCR在扩增子片段上引入通用接头,酶切产生粘性末端再连接成长序列,经过多次酶切-连接循环后形成包含约12~15个随机单体的串联子(concatemer)。对于短文库序列,纳米孔测序没有足够的信息进行基线信号均一化,因此,SAL不仅提高了测序通量,同时还改善了测序reads的质量。
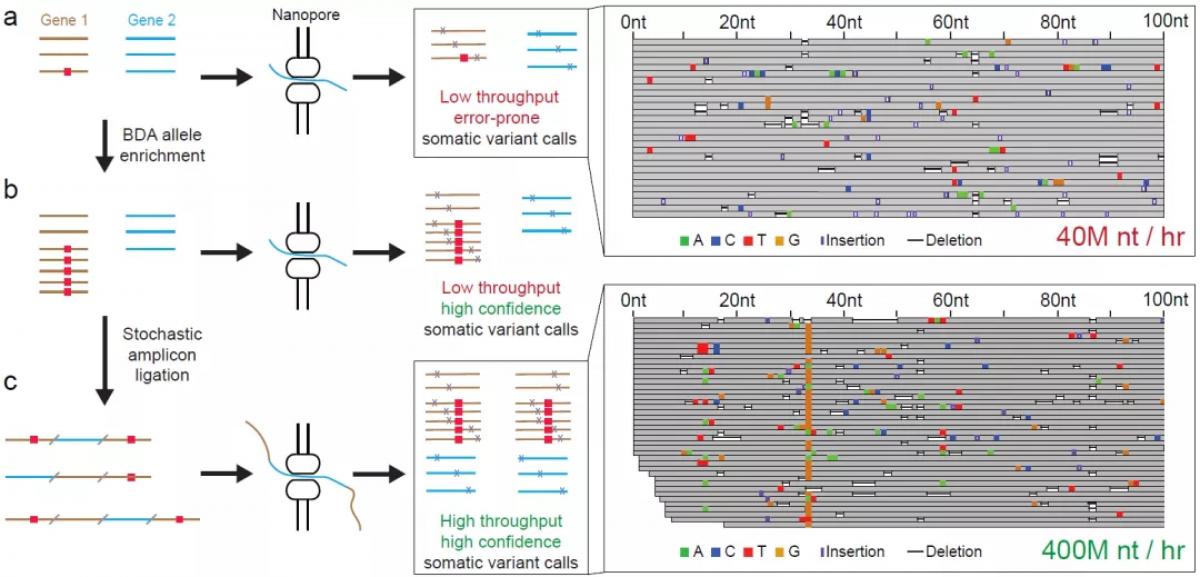
图2.
通过随机扩增子连接(SAL)对短扩增子进行串联。
想要达到5% VAF的检测限,SAL还需要同时检测配对的正常组织样本,这在临床上经常是比较难获取的。因此,要解决这个问题需要从样品起始端放大变异信号,BDA技术的引入恰好可以很好地解决上述问题。在多轮PCR过程中,特殊设计的blocker探针大大降低野生型DNA序列的扩增效率,使得突变型序列在产物中被富集出来。至此,完整的OCEANS技术建库流程包括:起始DNA样本通过BDA技术扩增并富集目标变异序列,产物经过SAL串联成长序列,筛选合适的片段大小并连接测序接头。整个过程包括最终的上机测序及数据分析步骤只需要大约10个小时,并且无需额外的配对样本。
结果显示,0.1% VAF的模拟样本产出了超过70%的VRF(Variant Read Fraction)信号。随后的panel实验表明,BDA技术在最终变异富集程度上起到决定性作用。
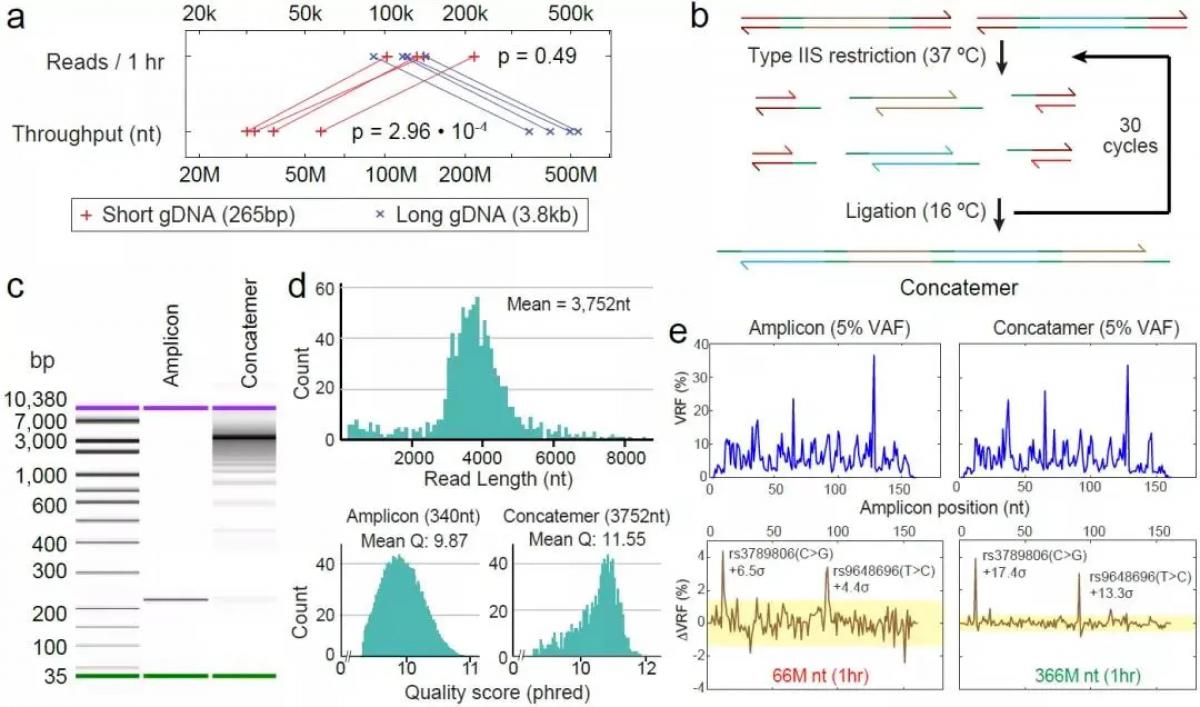
图3.
完整OCEANS技术建库流程及实验结果。
为测试OCEANS技术在多位点检测应用中的性能,研究团队分别构建了针对急性髓性白血病(AML)和黑色素瘤相关突变位点的小panel,每个panel覆盖大约300个变异位点。使用参考品测试的结果显示,最终检测限在0.05%~1%VAF之间。为了兼顾indel检测灵敏度和测序错误率的均一性,减少假阳性结果,研究团队在数据算法上也进行了优化。随后使用模拟样本(5%AML以及0.5%黑色素瘤)也都成功检测出预期突变,并且额外检出了一些真实存在的并且不包含在panel中的变异。
随后,研究团队利用黑色素瘤panel进行了真实临床样本的验证实验。根据之前性能确认的结果,OCEANS试验组的阳性阈值设定为20%VRF,NGS阈值设定为5% VAF,对全部9600个位点统计两种方法的一致性,灵敏度达到100%,特异性为99.0%,而OCEANS单独检出阳性的结果很可能也是真阳性。针对部分5%NGS VRF cutoff阈值以下的样本使用ddPCR进行验证,也获得了与OCEANS非常高的一致性,标志着OCEANS有潜力应用于微小病灶残留(MRD)检测的场景。当然,尽管我们使用了FFPE损伤修复试剂盒,检测结果很难完全排除体外DNA损伤导致的FFPE样本突变。
随后,研发人员设计了两个更大型的非小细胞肺癌(NSCLC)和肝癌(HCC)测序panel,与NGS方法相比,在实际样本上的一致性同样非常高。进一步分析OCEANS显著检出NGS的VRF为0%的位点,发现这些位点的测序深度普遍偏低(<300x)。
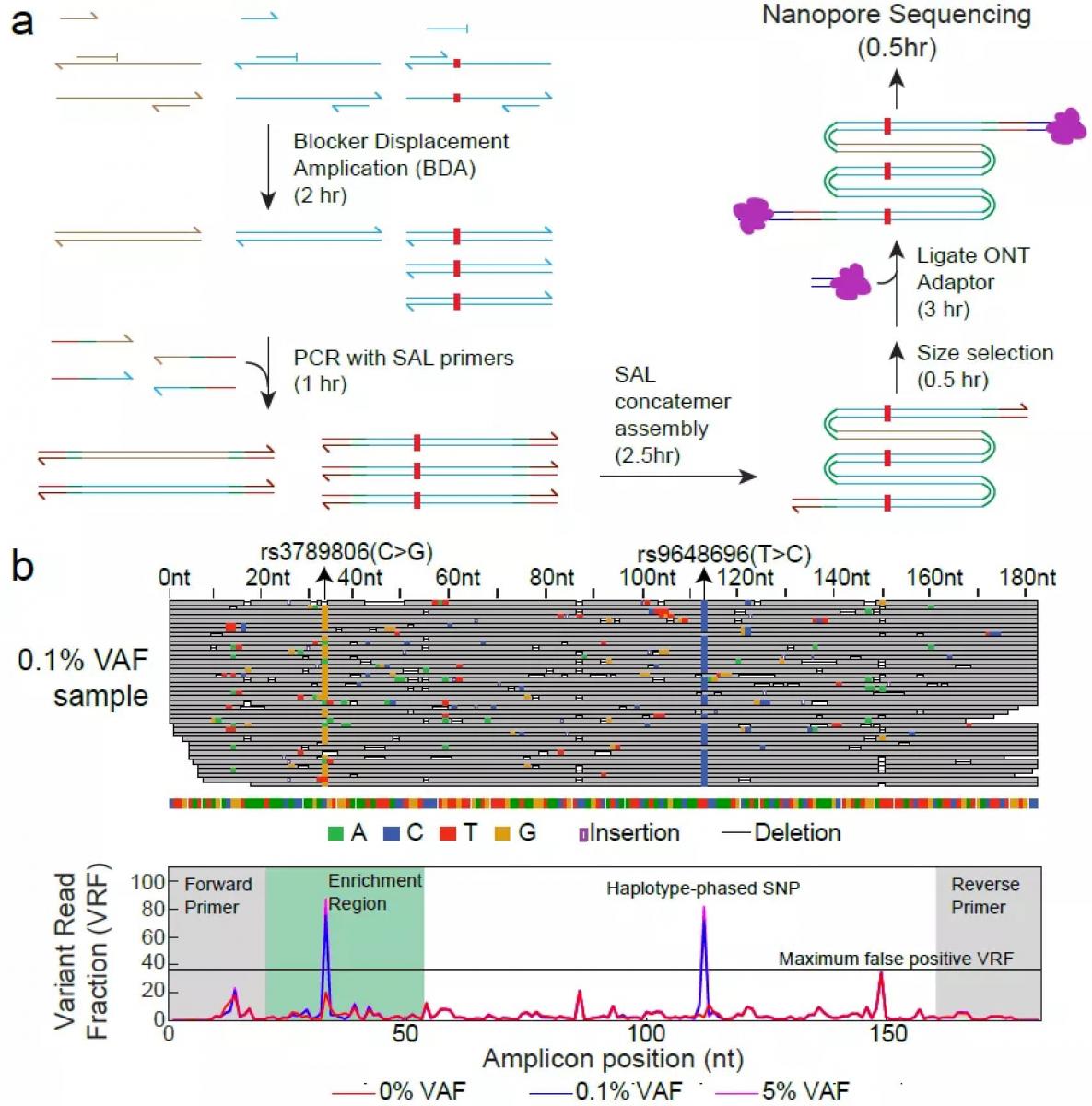
图4.
多基因OCEANS panels的生物学信息和somatic mutation calls检测限。
总之,OCEANS技术通过选择性富集体细胞中的突变序列并连接成长片段,使得Nanopore平台可以轻松检出目标区段的低频变异,检测限可达0.05%~1% VAF。在使用临床FFPE样本比较OCEANS与主流NGS方法的一致性实验中,AUC(Area Under the Curve)区域达到99.79%~99.99%,证明二者超高的一致性。同时,ddPCR也验证了OCEANS技术在低VAF水平(0.02%~0.66%)的检测可靠性,提示OCEANS技术在MRD监测中的应用潜力。
除了高灵敏度检测SNV的能力,借助纳米孔平台天然的快速、低成本优势,当日拿到panel测序结果也将成为可能。此外,长读长搭配点突变检测能力的特点也为开发系统性肿瘤测序panel提供了思路。较容易获得长序列的样本(血液瘤,新鲜实体瘤等)还可以同时检测重要的结构变异。
不同测序方法比较
“当日可出具检测报告,这是纳米孔测序获得科研人员青睐的重要原因,可以帮助临床更快地实施诊断和治疗。我们一直寻求在特定临床情景中应用纳米孔测序的方法,而针对癌症患者的敏感突变检测是目前关注的重点,因为速度在其中很关键。”该研究的通讯作者、MD Anderson癌症研究中心助理教授Lawrence Kwong表示,“OCEANS技术的建立赋予纳米孔测序高灵敏度突变检测的能力,为开发单日检测周期的测序panel蹚平了道路,最终的目标是使临床患者获益。”
当前,NGS可以很出色地进行高通量高灵敏度变异检测,OCEANS技术带给纳米孔测序类似能力的同时,也带来了更短的检测周期、更低的测序成本以及更长的读长等优势。通过真实设计的4种OCEANS panel,研究团队证明了该技术在临床应用中的巨大潜力。
“使用肿瘤FFPE临床样本进行的验证实验中,OCEANS与NGS技术的一致性超过了99%,拿到结果的时间还更快。”阅尔基因研究员、该文章第一作者Deepak Thirunavukarasu博士说,“据我们了解,这是首次报道在纳米孔测序平台实现对体细胞低丰度点突变的快速检测。”
“检测的准确性和时效性对临床决策都很重要,”该研究的共同通讯作者,阅尔基因美国创新中心负责人、莱斯大学生物工程终身教授David Zhang说, “阅尔基因致力于新技术的开发与转化,在精准医疗领域推出高灵敏度且周期短的检测产品。”
来源:测序中国